Se si naviga sul web si trova un po’ di tutto sull’opportunità o meno di consumare latte, sui benefici nutrizionali, sui rischi di contrarre malattie e sul fatto che l’uomo sia l’unico mammifero che consuma questo alimento da adulto, una cosa considerata “non naturale”. Molte volte le considerazioni sono basate su opinioni più che su dati scientifici.
Anche la ricerca scientifica non è unanime, ed i risultati di studi epidemiologici e nutrizionali sono a volte contradditori, probabilmente a causa di variabili difficili da controllare e che non dipendono dal consumo di latte. Per esempio, se in alcuni articoli viene riportato un aumento del rischio di tumori (Melnik, 2021) dovuto al consumo di latte, in altri il latte risulta avere un effetto protettivo (Vieira et al., 2017). Probabilmente la verità sta nel mezzo, cioè il consumo di latte non ha effetti rilevanti sul rischio di contrarre questa grave malattia ed i risultati diversi sono dovuti ad altri fattori, non facilmente identificabili. Ovviamente le cose cambiano nel caso in cui si sia allergici a qualche proteina del latte o intolleranti al lattosio. In questo caso per evitare l’instaurarsi di stati infiammatori, causa di aumento di rischio di diverse patologie, ci si dovrebbe astenere dal bere latte, oppure si dovrebbe consumare latte speciale, per esempio latte ad alta digeribilità nel quale il lattosio sia già “digerito” e scisso nei componenti glucosio e galattosio, o accontentarsi di consumare formaggio che già dopo poche settimane di stagionatura non contiene che tracce di lattosio.
Possiamo chiederci se esista un giudice che non si lascia influenzare dalle opinioni dei singoli o dall’altalenarsi dei risultati sperimentali, e che può darci un parere sull’opportunità o meno di bere latte. Il giudice imparziale c’è, ed è il nostro genoma. Con le nuove tecnologie di analisi genomica è possibile analizzarlo con grande dettaglio, grazie alle informazioni raccolte da centinaia di migliaia di marcatori del DNA. Studiandolo abbiamo imparato a distinguere i geni utili, selezionati in modo positivo dalla selezione naturale o artificiale, da quelli non selezionati e da quelli deleteri, selezionati in modo negativo. Possiamo quindi chiederci, e chiedere al nostro genoma, se il gene che ci permette di digerire il lattosio da adulti sia utile oppure no. Potremmo concludere che il latte faccia bene solo nel caso in cui questo gene porti segnali di una selezione positiva. Ma come è possibile capirlo?
La selezione lascia segni riconoscibili nel genoma delle popolazioni che ne sono state soggette. I segni della selezione possono essere rilevati attraverso l’analisi della variazione genetica e della distribuzione delle frequenze alleliche tra le popolazioni. Uno dei metodi per identificare regioni del genoma sotto selezione positiva utilizza la lunghezza degli aplotipi. Un aplotipo è una combinazione specifica di alleli su un cromosoma che è ereditata insieme, come un blocco unico. Nelle popolazioni gli aplotipi tendono ad accorciarsi a causa della ricombinazione che avviene durante la riproduzione sessuale. Durante la formazione degli ovuli e degli spermatozoi, i cromosomi omologhi si appaiano e scambiano segmenti di DNA attraverso un processo chiamato ricombinazione meiotica. Questo processo di ricombinazione genera nuove combinazioni di varianti genetiche che possono essere diverse da quelle presenti nei genitori. Contemporaneamente gli alleli dell’aplotipo originale possono ricombinare, formando aplotipi più corti (Figura 1).
Figura 1. La lunghezza di un aplotipo (verde) diminuisce nel corso delle generazioni a causa della ricombinazione.
Aplotipi sotto selezione positiva aumentano di frequenza in poche generazioni nella popolazione (Figura 2), quindi ricombinano meno e sono decisamente più lunghi rispetto ad aplotipi che non sono sotto selezione positiva.
Figura 2. Mutazioni favorevoli aumentano di frequenza in poche generazioni a causa della selezione positiva. Di conseguenza ricombinano meno volte rispetto ad alleli non sotto selezione.
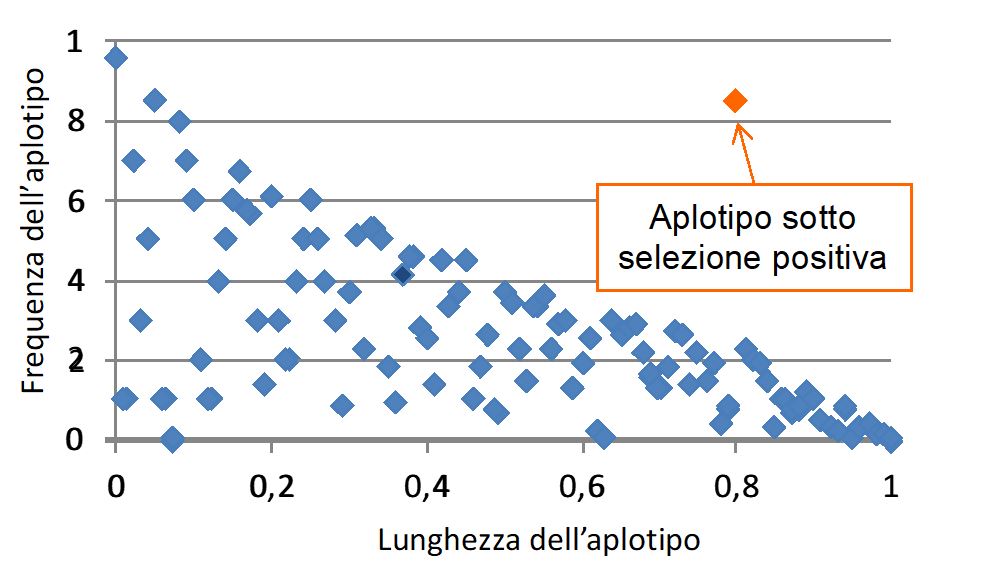
La frequenza e la lunghezza di un aplotipo può essere quindi utilizzata per valutare la forza della selezione naturale che agisce su una popolazione (Figura 3).
Figura 3. Gli aplotipi che comprendono mutazioni favorevoli sono frequenti e di dimensioni maggiori rispetto agli altri.
Che storia racconta il nostro genoma sul gene della lattasi?
La lattasi è l’enzima prodotto nell’intestino tenue che è responsabile della digestione del lattosio, lo zucchero del latte. Nella maggior parte dei mammiferi, incluso l’uomo, l’espressione del gene lattasi è regolata durante la crescita e cessa dopo lo svezzamento. Questo significa che gli adulti di solito non producono lattasi e non sono in grado di digerire il lattosio. Tuttavia, in alcune popolazioni umane, la produzione di lattasi non diminuisce dopo lo svezzamento, ma continua per tutta la vita.
Il gene della lattasi (LCT) ha varianti dovute a mutazioni diverse avvenute nel tempo ed in popolazioni diverse. Per esempio, negli europei la mutazione che causa la persistenza della produzione di lattasi è stata identificata in un singolo nucleotide del gene LCT, che codifica per l’enzima lattasi (Itam et al., 2009). Questa mutazione non era presente nei primi neolitici europei ed è emersa circa 7.500 anni fa (Burger et al., 2007). È nota come la mutazione C/T – 13910 e si trova vicino al promotore del gene LCT che regola l’espressione del gene e quindi la produzione di lattasi. Altre mutazioni che inducono sono state osservate in Africa Medio Oriente e in Asia (Figura 4; Ingram et al., 2009; Ranciaro et al., 2014; Mortinaro et al., 2021).
Figura 4. Mappa della distribuzione mondiale di diversi varianti del gene della lattasi che ne determinano la persistenza durante l’età adulta (tratto da Anguita-Ruiz et al., 2020).
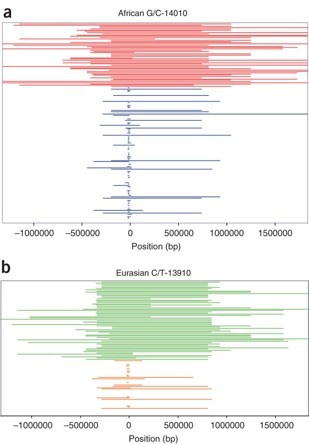
Queste mutazioni hanno chiari segni di selezione positiva. Alcune sono molto frequenti nelle popolazioni nelle quali sono avvenute, e tutte sono localizzate in aplotipi molto più lunghi rispetto agli aplotipi omologhi che non portano le mutazioni che determinano la persistenza della lattasi (Figura 5; Bersaglieri et al., 2004 Tishkoff et al., 2006).
Figura 5. Lunghezza degli aplotipi in corrispondenza del gene LCT (da Tishkoff et al. 2006). Gli aplotipi che portano la mutazione per la persistenza da adulti (in rosso la mutazione africana G/C-14010 e in verde la mutazione europea C/T-13910) sono molto più lunghi degli aplotipi omologhi che non portano la mutazione (in blu allele africano non persistente e in arancione allele europeo non persistente).
In generale, la frequenza del gene della lattasi è più alta nelle popolazioni che hanno tradizionalmente praticato l’allevamento del bestiame come fonte di cibo, come le popolazioni europee, africane orientali e meridionali, mediorientali e asiatiche meridionali (Figura 6; Segurel et al., 2017; Anguita-Ruiz et al., 2020).
Figura 6. Frequenza della persistenza della lattasi nel mondo (tratto da Anguita-Ruiz et al., 2020).
In queste popolazioni, la capacità di digerire il lattosio nell’età adulta è stata vantaggiosa, poiché il latte e i suoi derivati sono fonti ricche di proteine e nutrienti importanti. Ciò ha permesso alle popolazioni che hanno mantenuto l’espressione del gene lattasi di espandersi e prosperare in regioni dove altre fonti di cibo erano limitate. Inoltre, ha portato allo sviluppo di culture e di pratiche alimentari basate sul consumo di prodotti lattiero-caseari.
D’altra parte, la frequenza del gene della lattasi è generalmente più bassa nelle popolazioni che non hanno avuto una tradizione di allevamento del bestiame, come le popolazioni asiatiche orientali, le popolazioni native americane e molte popolazioni africane occidentali.
La frequenza del gene della lattasi sta cambiando in molte popolazioni in tutto il mondo a causa di fattori come la migrazione, l’urbanizzazione e l’adozione di pratiche alimentari diverse.
In conclusione, per chi ha la mutazione giusta, non c’è dubbio, il latte fa bene!
Ringraziamenti
Si ringrazia la Fondazione Romeo ed Enrica Invernizzi per il finanziamento al Centro di Referenza IRCAF
Autori
Paolo Ajmone Marsan, Simone Morabito e Giuseppe Bertoni – Dipartimento di Scienze Animali, della Nutrizione e degli Alimenti – DIANA e Centro di riferimento agro-alimentare
Romeo ed Enrica Invernizzi – IRCAF, Università Cattolica del Sacro Cuore
Referenze
Anguita-Ruiz A, Aguilera CM, Gil Á. Genetics of Lactose Intolerance: An Updated Review and Online Interactive World Maps of Phenotype and Genotype Frequencies. Nutrients. 2020 Sep 3;12(9):2689. doi: 10.3390/nu12092689. PMID: 32899182; PMCID: PMC7551416.
Bersaglieri T, Sabeti PC, Patterson N, Vanderploeg T, Schaffner SF, Drake JA, Rhodes M, Reich DE, Hirschhorn JN. Genetic signatures of strong recent positive selection at the lactase gene. Am J Hum Genet. 2004 May;74(5):1111-20. doi: 10.1086/421051. PMID: 15042509; PMCID: PMC1181964.
Burger J, Kirchner M, Bramanti B, Haak W, Thomas MG. Absence of the lactase-persistence-associated allele in early Neolithic Europeans. PNAS March 6, 2007 vol. 104 no. 10, 3736–3741. www.pnas.org cgi doi 10.1073 pnas.0607187104.
Ingram CJ, Mulcare CA, Itan Y, Thomas MG, Swallow DM. Lactose digestion and the evolutionary genetics of lactase persistence. Hum Genet. 2009 Aug;124(6):579-91. doi: 10.1007/s00439-008-0593-6. Epub 2008 Nov 12. PMID: 19002438; PMCID: PMC2711582.
Itan Y, Powell A, Beaumont MA, Burger J, Thomas MG. The origins of lactase persistence in Europe. PLoS Comput Biol. 2009 Aug;5(8):e1000491. doi: 10.1371/journal.pcbi.1000491. Epub 2009 Aug 14. PMID: 19680426; PMCID: PMC2722739.
Melnik BC. Lifetime Impact of Cow’s Milk on Overactivation of mTORC1: From Fetal to Childhood Overgrowth, Acne, Diabetes, Cancers, and Neurodegeneration. Biomolecules. 2021 Mar 9;11(3):404. doi: 10.3390/biom11030404. PMID: 33803410; PMCID: PMC8000710.
Montinaro F, Busby GB, Gonzalez-Santos M, Oosthuitzen O, Oosthuitzen E, Anagnostou P, Destro Bisol G, Drayson MT, Raza MW, Fugman DA, Spector TD, Steves CJ. A timeline of lactase persistence in Africa: implications for colonialism and the slave trade. BMC Evol Biol. 2021 Jun 25;21(1):105. doi: 10.1186/s12862-021-01844-6. PMID: 34171994; PMCID: PMC8222482.
Ranciaro A, Campbell MC, Hirbo J, Thompson S, Jojohunnes Y, Mohamed H, Lu TT, Woldemeskel D, Froment A, Omar SA, Bodo JM, Nyambo T, Belay G, Drayna D, Bishara A, Okeyo-Owuor J, Ranciaro A, Hirbo J, Thompson S, Omar SA, Drayna D, Belay G, Froment A, Nyambo T, Bodo JM, Woldemeskel D, Tishkoff SA. Genetic origins of lactase persistence and the spread of pastoralism in Africa. Am J Hum Genet. 2014 Feb 6;94(2):496-510. doi: 10.1016/j.ajhg.2014.01.019. PMID: 24508867; PMCID: PMC3928666.
Segurel L, Bon C. On the Evolution of Lactase Persistence in Humans. Annu. Rev. Genom. Hum. Genet. 2017. 18:297–319. https://doi.org/10.1146/annurev-genom-091416-035340.
Tishkoff SA, Reed FA, Ranciaro A, Voight BF, Babbitt CC, Silverman JS, Powell K, Mortensen HM, Hirbo JB, Osman M, Ibrahim M, Omar SA, Lema G, Nyambo TB, Ghori J, Bumpstead S, Pritchard JK, Wray GA, Deloukas P. Convergent adaptation of human lactase persistence in Africa and Europe. Nat Genet. 2007 Jan;39(1):31-40. doi: 10.1038/ng1946. Epub 2006 Dec 10. PMID: 17159977; PMCID: PMC2672153.
Vieira AR, Abar L, Chan DSM, Vingeliene S, Polemiti E, Stevens C, Greenwood D, Norat T. Foods and beverages and colorectal cancer risk: a systematic review and meta-analysis of cohort studies, an update of the evidence of the WCRF-AICR Continuous Update Project. Ann Oncol. 2017 Aug 1;28(8):1788-1802. doi: 10.1093/annonc/mdx171. PMID: 28407090.
(fonte Ruminantia)